来源公众号:生物奇迹biomiracle 作者:生物奇迹
限制性核酸内切酶(Restriction Endonuclease)是一类能识别双链 DNA 分子中特定核苷酸序列,并在特定位置切割 DNA 的酶,广泛存在于细菌和古菌中,是天然免疫系统的一部分,用于抵御外源 DNA(如噬菌体)的入侵。根据酶的结构、辅助因子需求、识别序列特征及切割位点的不同,可分为以下几种类型。
I 型限制性核酸内切酶
特点:
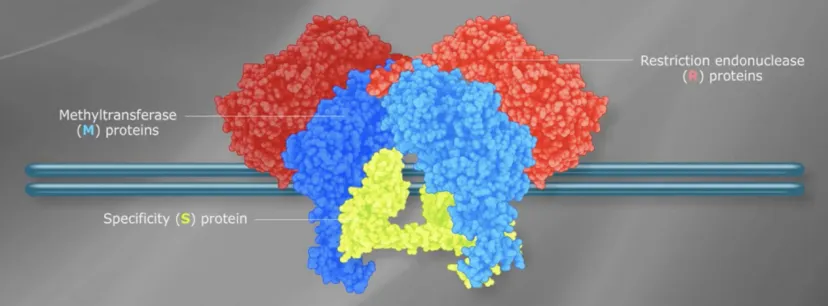
多亚基复合体:由修饰亚基(Mod)、限制亚基(Res)和识别亚基(HsdS)组成。
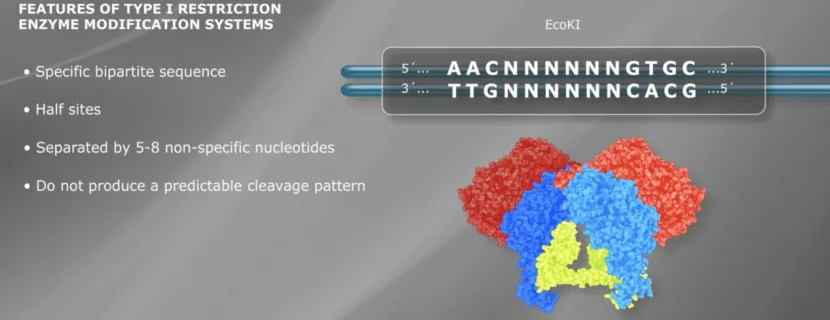
识别序列:通常为长序列(如EcoK的识别序列为 5′-AACNNNNNNGTGC-3’),包含特异性识别位点和非特异性间隔区域。
切割方式:切割位点远离识别序列(通常在识别位点下游 1000-2000 bp 处),切割位置随机,无固定末端。
辅助因子:需要 ATP、Mg²⁺和 S – 腺苷甲硫氨酸(SAM),切割过程消耗 ATP。
应用:因切割位置随机,难以用于基因工程,主要用于研究 DNA 转位和甲基化机制。
举例:
EcoK:识别序列为 5′-AACNNNNNNGTGC-3’。
EcoB:识别序列为 5′-TGANNNNNNNNTGCT-3’。
II 型限制性核酸内切酶
特点:
单亚基酶:结构简单,多数为同源二聚体。
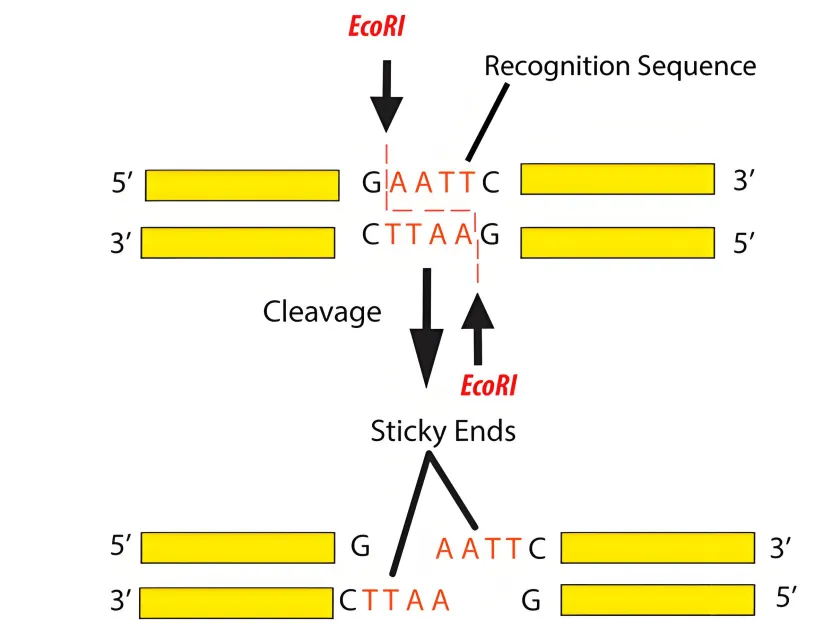
识别序列:多为 4-8 bp 的回文序列(如EcoRI的识别序列为 5′-GAATTC-3’),少数为非回文序列。
切割方式:直接在识别序列内或附近精确切割,产生粘性末端(如 5′ 突出或 3′ 突出)或平末端。
辅助因子:仅需 Mg²⁺,无需 ATP 或 SAM。
应用:基因工程中最常用的工具酶,用于 DNA 克隆、酶切图谱构建等。

举例:
1、产生粘性末端的酶:
EcoRI:识别 5′-GAATTC-3’,切割后产生 5′ 突出末端(G↓AATTC)。
HindIII:识别 5′-AAGCTT-3’,切割后产生 5′ 突出末端(A↓AGCTT)。
BamHI:识别 5′-GGATCC-3’,切割后产生 5′ 突出末端(G↓GATCC)。
2、产生平末端的酶:
SmaI:识别 5′-CCCGGG-3’,切割后产生平末端(CCC↓GGG)。
HaeIII:识别 5′-GGCC-3’,切割后产生平末端(GG↓CC)。
3、非回文序列识别酶:
BtgI:识别 5′-CACNNNGTG-3’,切割产生粘性末端。
III 型限制性核酸内切酶
特点:
异源二聚体:由 Mod(甲基化酶)和 Res(限制酶)亚基组成。
识别序列:由两个反向重复序列组成,间隔 1-2 bp(如EcoPI的识别序列为 5′-AGACC-3′ 和 5′-GTCTG-3’,间隔 1 bp)。
切割方式:在识别序列下游 24-26 bp 处切割,切割位点相对固定但不在识别序列内。
辅助因子:需要 ATP 和 Mg²⁺,但 ATP 仅用于激活酶,不被水解。
应用:较少用于基因工程,研究价值高于实用价值。
举例:
EcoPI:识别序列为 5′-AGACC-N-GTCTG-3’,切割位点在下游 25 bp 处。
EcoP15I:识别序列为 5′-CTGGA (N) 6-3’。
IV 型限制性核酸内切酶
特点:
识别修饰 DNA:专门作用于甲基化、羟甲基化或糖基化的 DNA,不识别特定核苷酸序列,而是识别 DNA 修饰状态。
切割方式:切割修饰后的 DNA 链,导致双链断裂。
辅助因子:依赖 Mg²⁺或 Mn²⁺。
应用:参与细菌对抗甲基化噬菌体 DNA 的防御机制,研究 DNA 修饰与表观遗传。
举例:
McrBC:识别甲基化的胞嘧啶(如 m⁵C),切割含甲基化 DNA 的区域。
Mrr:识别甲基化的腺嘌呤(如 m⁶A)或胞嘧啶,切割修饰后的 DNA。
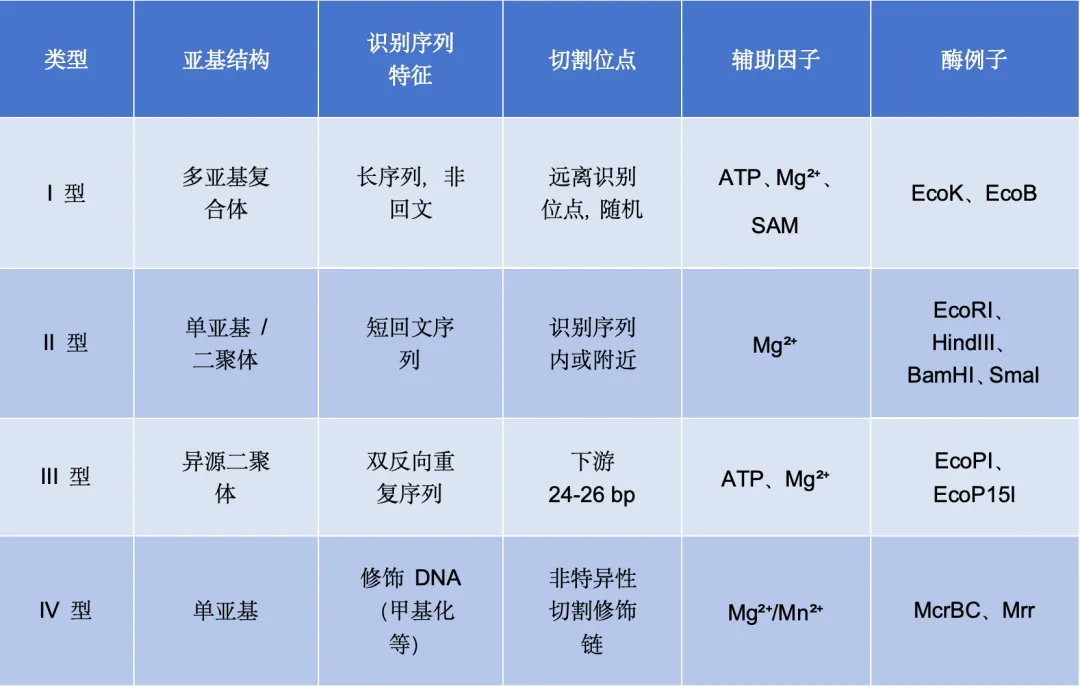 总结对比
总结对比
扩展知识
命名规则:通常以来源微生物属名首字母(大写)+ 种名前两个字母(小写)+ 菌株或血清型编号 + 罗马数字(如 EcoRI 来自Escherichia coli RY13 菌株,第 1 个发现的酶)。
星号标记:若酶识别序列中某个碱基被甲基化,可能改变切割特异性(如 EcoRI * 表示识别甲基化 DNA)。



 扫码安装网站APP(Android版)
扫码安装网站APP(Android版)


近期评论