来源公众号:生物帮SA
前期有朋友留言想要咨询关于质粒单一原件组成及构建方法,因此我们邀请生物帮SA成员撰稿。
无论是蛋白表达、基因敲除,几乎所有的分子实验都离不开质粒。看似复杂的质粒,其实是由一系列功能明确的元件组装而成。只要掌握这些“模块”的属性和组合逻辑,我们就可以根据自己的需求构建出对应的功能性质粒。本文将介绍质粒的重要原件组成以及质粒构建方法。
🧫单一原件质粒的单一元件指的是一个独立的功能模块。从下面这张质粒图可以看到比较重要的几个模块
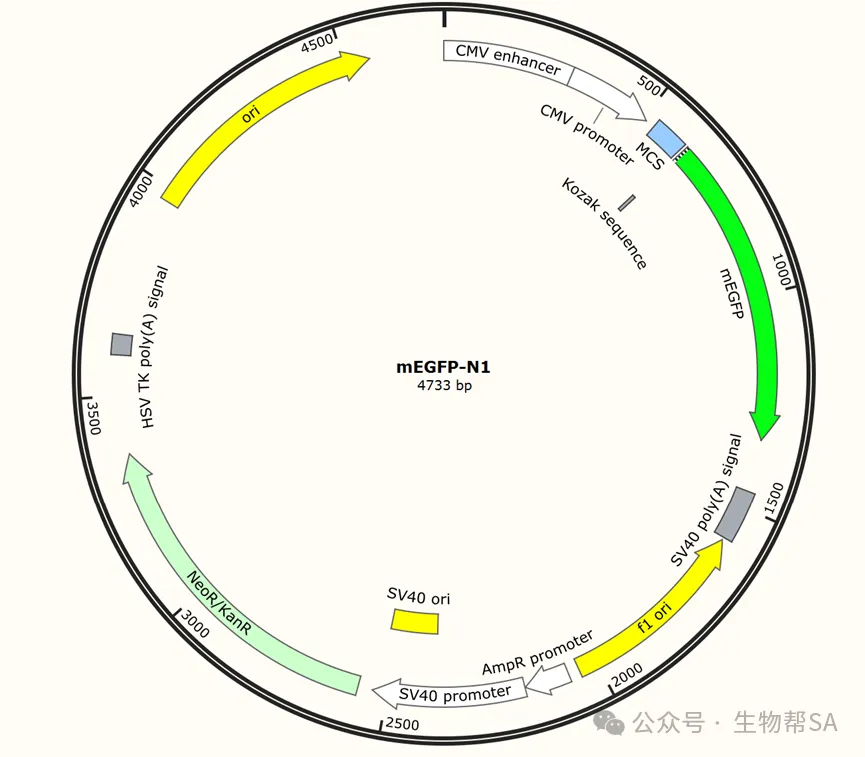
对于一个质粒,一般包括以下元件:
| 功能模块 | 作用说明 | 常见选择 |
| 复制起点(Ori) | 决定质粒在宿主细胞中的复制能力与拷贝数 | ColE1、pUC、p15A |
| 抗性基因 | 用于抗生素筛选阳性克隆 | AmpR、KanR |
| 启动子(Promoter) | 决定基因的表达时空与强度 | CMV、EF1α、T7、U6 |
| 编码序列(CDS) | 目标蛋白/功能RNA的表达框架 | GFP、Cas9、CARF |
| 标签(Tag) | 用于纯化、定位或免疫检测 | His、FLAG、HA、GFP |
| 终止子(Terminator) | 终止转录、增加mRNA稳定性 | SV40 polyA、BGH polyA |
| 多克隆位点(MCS) | 包含多个酶切位点,便于插入目标基因 | NdeI、AscI |
(1)复制起点(ori):控制质粒在宿主细胞(如大肠杆菌)中复制。例:ColE1(高拷贝数,适合克隆)或pSC101(低拷贝数,适合稳定遗传)。
| Ori 名称 | 宿主类型 | 拷贝数 | 特点 |
| ColE1 | 大肠杆菌 | 中高拷贝 | 最常用,兼容 pUC 系列 |
| pUC ori | 大肠杆菌 | 超高拷贝 | 改造自 ColE1,表达量高 |
| p15A | 大肠杆菌 | 低拷贝 | 与 ColE1 兼容,可双质粒共转 |
| SV40 Ori | 哺乳动物 | 中拷贝 | 需 SV40 T 抗原辅助复制 |
| oriP/EBNA1 | 哺乳动物 | 稳定拷贝 | 在含 EBNA1 蛋白的细胞中维持复制 |
(2)抗性基因:用于筛选含质粒的细胞,通常是抗生素抗性基因,如氨苄青霉素抗性基因(ampR)或卡那霉素抗性基因(kanR)。
(3)启动子:控制目标基因转录,不同表达系统中启动子的选择是不同的。
| 启动子 | 类型 | 应用场景 | 特点 |
| CMV | 哺乳动物强启动子 | 过表达、瞬时转染 | 表达强,广谱 |
| EF1α | 哺乳动物中强启动子 | 稳定表达系统 | 活性稳定,不易沉默 |
| SV40 | 哺乳动物 | 报告基因驱动 | 中等强度 |
| U6 / H1 | RNA Pol III 启动子 | sgRNA/shRNA 表达 | 控制非翻译RNA |
| T7 | 原核表达(体外转录) | 体外表达系统 | 需T7 RNA聚合酶 |
| lac / trc / tac | 可诱导原核启动子 | IPTG 诱导表达蛋白 | 适合大肠杆菌表达蛋白 |
(4)目标基因/标签:该模块用于编码目标蛋白/标签。
(5)终止子:终止子是基因末端的DNA序列,负责终止转录并确保RNA分子正确加工。
(6)多克隆位点(Multiple Cloning Site,MCS):MCS区域包含多种限制性酶切位点,通过酶切和重组引入外源基因。
🧬 构建功能质粒的模块化思维
一个典型的质粒可以分为骨架模块和表达模块两大部分:
1. 骨架模块(Backbone)
这是所有质粒的“基础设施”,确保质粒能在宿主中复制、筛选。通常包括:复制起点(Ori);抗生素抗性基因(如 AmpR)
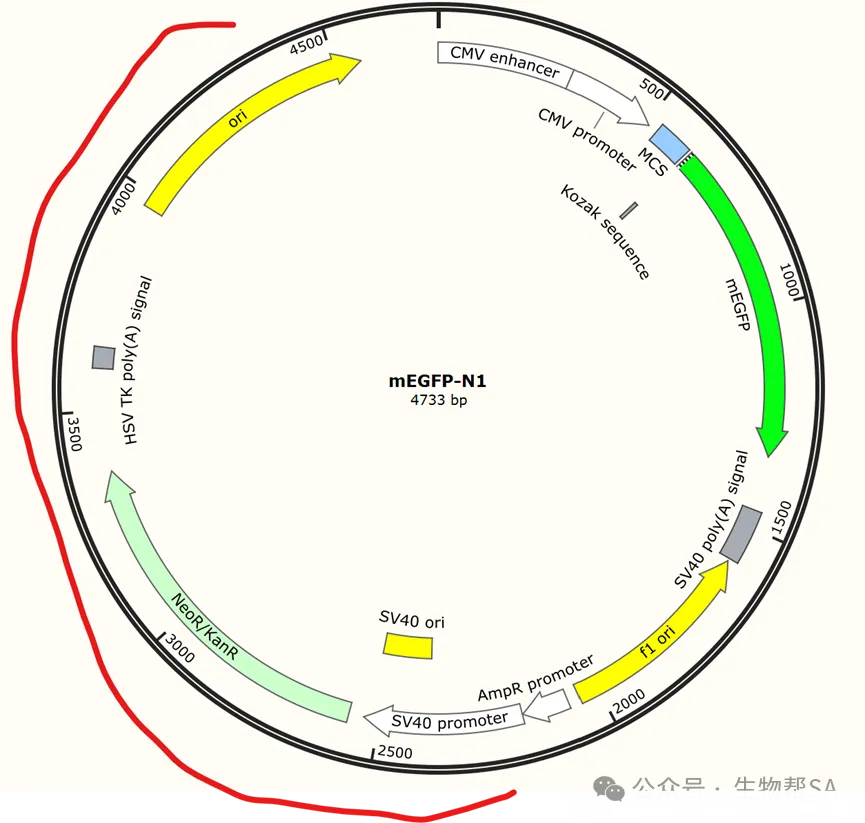
2. 表达模块(Expression Cassette)
这是质粒实现功能的“工作单元”,其结构一般为:[启动子] – [目标基因] – [标签] – [终止子]
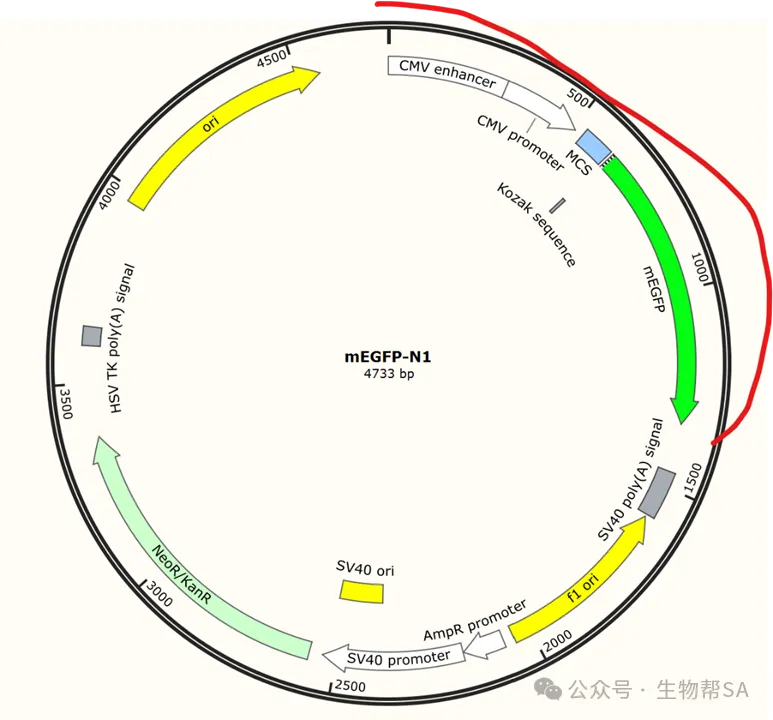
另外根据个人需求可以加入个性化序列,例如这里的f1 ori使质粒在 M13 感染细胞中生成单链 DNA,便于测序或突变。
🧫构建功能质粒的步骤
1. 定义研究目的
明确这个质粒的功能——表达蛋白?RNA干扰?基因编辑?抗性筛选?
2. 挑选功能元件
根据实验目的以及各个元件的功能进行选择,各个元件的功能及应用场景见上面的内容。
3. 设计连接方式
(1)限制性内切酶 + T4 DNA连接酶
🔹原理
利用内切酶识别特定位点切割质粒和目的片段,使之产生互补的“粘性末端”或“平末端”,再通过连接酶(T4 DNA ligase)将片段与载体连接。
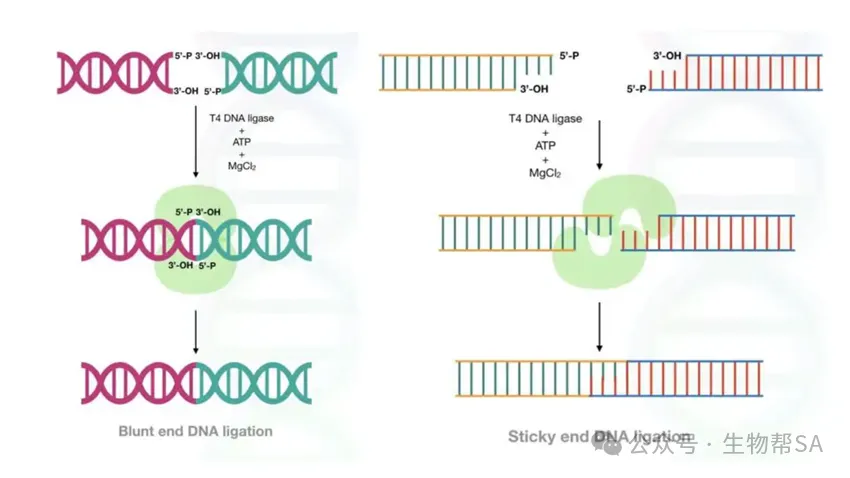
T4(Genetic Education)
🔹优点
·技术成熟、操作简便
·实验成本低
·适合初学者或经典构建任务
🔹缺点
·需确保没有干扰性限制酶位点
·多片段连接困难
·通常会在连接处留下“酶位点痕迹”
🔹适用场景
构建简单表达载体(如插入一个目的基因)
(2)Gibson Assembly(无缝连接法)
🔹原理
将具有重叠序列(15–40 bp)的片段在等温反应中用 5′ 外切酶、聚合酶和连接酶处理,实现片段的无缝拼接。
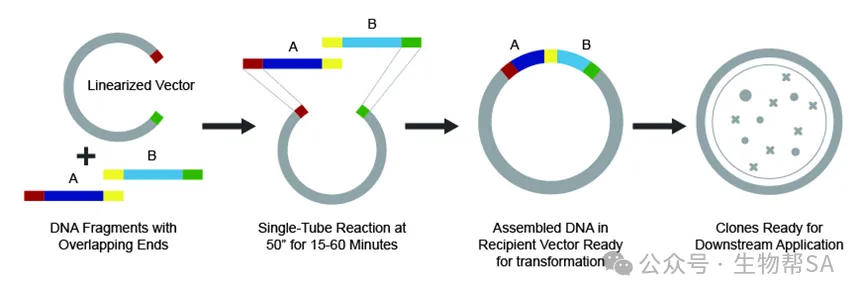
Gibson Assembly (Molecularcloud)
🔹优点
·无需限制酶,连接处无痕
·支持多片段一次性连接
·整个反应只需一个管、一个步骤
🔹缺点
要求精确设计重叠区
对 DNA 片段纯度、量要求高
成本略高于传统方法
🔹适用场景
大于2个片段拼接;构建复杂表达盒;CRISPR载体拼接等
(3)Golden Gate Assembly
🔹原理
使用 Type IIS 限制酶(如 BsaI、BsmBI)识别位点切割位点在识别序列外侧,可通过设计使连接片段按顺序无缝拼接。

Golden Gate Assembly(SnapGene)
🔹优点
·支持模块化、标准化构建
·单管反应,效率高
·无缝拼接,连接顺序可控
🔹缺点
·对 Type IIS 酶识别位点敏感,需避免片段内含有相应位点
·初次设计较复杂
🔹适用场景
高通量克隆、合成生物学、植物载体构建、模块化表达系统
(4)In-Fusion Cloning(同源重组型克隆)
🔹原理
利用 15 bp 左右的同源臂,通过专用酶(如Clontech 的In-Fusion 酶)将 PCR 产物直接插入线性化载体。
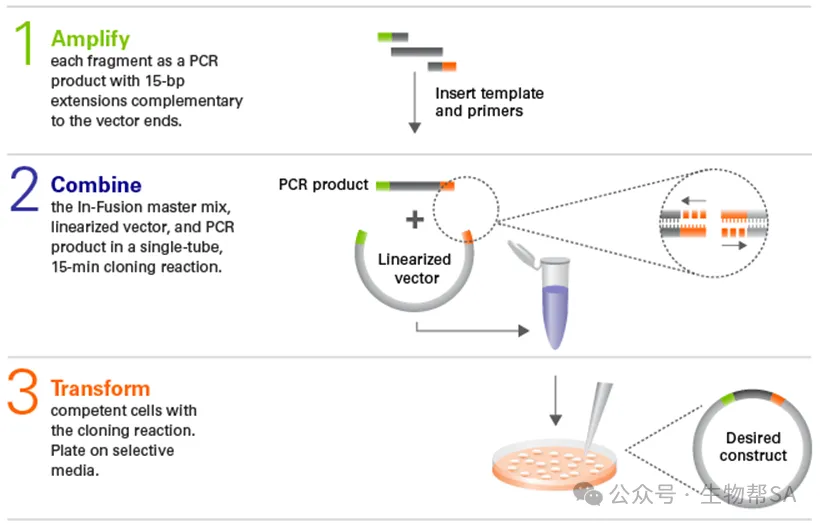
In-Fusion Cloning(Takara Bio)
🔹优点
· 无缝连接、无需限制酶
· 操作快速、结果可靠
· 可连接多个片段
🔹缺点
· 需使用专用试剂盒,成本较高
· 连接效率依赖于同源臂设计质量
🔹适用场景
快速插入表达框;构建融合蛋白;需要精确无痕插入的项目
4. 模拟质粒结构
使用软件如SnapGene,将每一个模块按顺序拼接起来,并检查:阅读框是否连贯;限制酶位点是否重复;元件间有无功能冲突;有无不必要的重复序列
来源网址:质粒元件组成及构建方法



 扫码安装网站APP(Android版)
扫码安装网站APP(Android版)


近期评论