来源公众号:生物侯老师
一、质粒图谱概述
质粒图谱是环状 DNA 分子的物理信息可视化展示,包含复制原点、筛选标记、多克隆位点、表达调控元件等关键信息。作为基因克隆与表达的重要工具,图谱解析是载体选择与实验设计的基础。
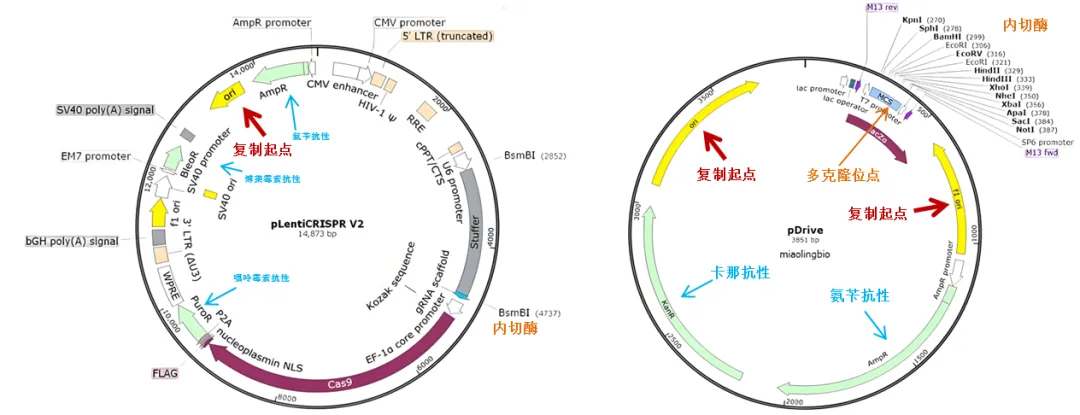
二、核心组成要素
- 复制原点(Ori)
- 原核质粒:单复制起点(如 pMB1)
- 真核质粒:含 SV40 等真核复制信号
- 穿梭质粒:双复制起点(如 ColE1+SV40)
- 筛选标记系统
- 抗生素抗性基因:Ampⁿ(氨苄)、Kanⁿ(卡那)、Neoⁿ(新霉素)
- 营养缺陷型标记:Leu⁺、Trp⁺等(酵母载体常用)
- 多克隆位点(MCS)
- 特征:密集排列的单一酶切位点(如 EcoRI、HindIII)
- 功能:外源基因插入窗口,通常位于启动子下游
- 表达调控元件
- 启动子:CMV(强)、T7(原核)、U6(RNAi)
- 增强子 / 沉默子:位置不固定的转录调控序列
- 终止子:polyA 信号(真核)、rho-independent(原核)
三、图谱解析要点
- 方向性识别
- 转录方向:启动子箭头(白色)指向基因编码区
- 复制方向:Ori 箭头(黄色)指示 DNA 合成方向
- 链区分:顺时针箭头对应正链(编码链),逆时针对应负链
- 载体类型判断
- 原核载体:单一 Ori + LacZα 筛选系统
- 真核载体:含 Kozak 序列或内含子剪接信号
- 病毒载体:含 LTR、包装信号等特征序列
- 克隆可行性评估
- 容量限制:常规质粒 < 10kb,BAC 可达 300kb
- 酶切策略:选择 MCS 中未被其他元件占用的酶切位点
- 表达效率:强启动子(如 EF1α)搭配优化的 UTR 序列
四、高级功能模块
- 融合标签系统
- N 端标签:His₆、GST(促进蛋白纯化)
- C 端标签:Flag、HA(利于免疫检测)
- 双向标签:IRES 连接双基因表达
- 特殊功能元件
- 荧光报告基因:EGFP、mCherry
- 重组酶位点:loxP、FRT
- 复制调控系统:严谨型(pSC101)/ 松弛型(pUC)
五、应用注意事项
- 转化筛选时需匹配正确抗生素浓度
- 大片段克隆建议使用低拷贝载体(如 pBR322)
- 真核表达需验证 Kozak 序列完整性
- 注意载体与宿主菌的基因型匹配(如 dam/dcm 甲基化)
六、图谱阅读技巧
- 顺时针 / 逆时针箭头:表示不同 DNA 链的转录方向
- 颜色编码:常用白色(启动子)、黄色(Ori)、蓝色(抗性基因)
- 分子量标注:图谱边缘的数字代表 kbp 长度
七、高频考点
- 限制酶选择与重组质粒构建
- 双酶切优势使用两种不同限制酶切割质粒和目的基因,可避免质粒自连并确保插入方向正确。
- 酶切位点选择优先选择 MCS 中未被其他元件(如启动子、抗性基因)占用的位点,同时考虑酶切后产生的黏性末端是否匹配。
- 易错点若目的基因内部含所选限制酶的识别序列,则需调整酶切方案。
- 筛选与检测方法
- 抗生素筛选根据质粒抗性基因选择对应抗生素(如 Ampⁿ用氨苄青霉素),重组质粒若插入外源基因导致抗性基因失活,则无法在含该抗生素的培养基上生长。
- 蓝白斑筛选利用 LacZα 基因(编码 β- 半乳糖苷酶),插入外源基因会破坏该基因,导致菌落呈白色(非重组菌落为蓝色)。
- 分子检测PCR 验证(引物设计需跨插入位点)、酶切鉴定(双酶切后电泳观察片段大小)、测序确认。
- 实验设计与结果分析
- 目的基因表达验证通过 Western blot 检测目的蛋白,或通过荧光报告基因(如 EGFP)观察表达情况。
- 对照实验设置需设置阳性对照(含已知目的基因的质粒)和阴性对照(未转化的宿主菌)。
来源网址:【精准备考】如何读懂质粒图谱及高频考点



 扫码安装网站APP(Android版)
扫码安装网站APP(Android版)



近期评论